大家好!今天小编给大家分享的是一篇于2023年4月同我司合作发表在Frontiers in Immunology杂志(IF: 8.786)的文章《Characterization of the fatty acid metabolism in colorectal cancer to guide clinical therapy》。
背景介绍
肝纤维化(HF)是由各种慢性肝病如病毒性肝炎、酒精性肝炎、胆汁淤积症和药物性肝损伤等引起的过度修复反应所导致的异常细胞外基质沉积。HF已成为全球一个日益严重的公共健康问题,可能导致肝硬化和肝癌等高危严重肝病的发生。然而,目前还没有任何HF治疗药物获临床批准使用。因此,探索HF的发病机制对于发现肝纤维化的靶向治疗药物具有重要意义。HF的直接原因是过量生成和沉积胶原、糖蛋白和蛋白多糖等细胞外基质。HF的发生涉及多个细胞变化。肝星状细胞激活和向肌成纤维细胞转化是HF发展的中心环节。激活的肝星状细胞通过产生大量细胞外基质导致HF的形成。HF过程中常伴随着肝脏炎症损伤。肝脏炎症和肝脏免疫微环境改变被认为是HF发展的关键因素。肝脏含有丰富的免疫细胞,包括先天免疫细胞(库普弗细胞、自然杀伤细胞、自然杀伤T细胞)和获得性免疫细胞(T细胞和B细胞)。越来越多的研究表明,免疫细胞调节HF的进展和消退。在HF过程中,免疫系统通过触发炎症参与组织损伤的修复。肝脏损伤后,肝脏中的免疫细胞被激活并招募到损伤部位,通过分泌肿瘤坏死因子α(TNF-α)、白细胞介素-6(IL-6)、白细胞介素-1β(IL-1β)等促炎细胞因子来激活HSC或损伤的肝细胞。免疫相关基因和信号通路在HF肝组织的免疫细胞浸润中起重要作用。
近年来,随着测序技术的发展,生物信息学已成为广泛用于疾病生物标志物、病理机制和潜在治疗药物识别的良好分析方法。本研究整合生物信息学、转录组学和动物实验,识别了与HF相关的免疫基因和生物通路,有助于我们进一步理解HF的发病机制,为HF的诊断和治疗做出贡献。
全文速览
肝纤维化(HF)的发生和发展伴随着炎症性损害。免疫基因通过调节免疫细胞的浸润,在HF的纤维生成和炎症损害中起着关键作用。然而,对HF的免疫机制的研究还不够充分。因此,本研究旨在确定参与HF纤维化形成和炎症性损害的免疫基因和生物途径,并探索基于免疫靶点的HF治疗方法。研究者从GEO数据库下载HF的表达数据集GSE84044。根据加权基因共表达网络分析(WGCNA)筛选出HF的关键模块基因。将关键模块基因与ImmPort数据库中的免疫相关基因进行比对,得到肝纤维化免疫基因(HFIGs)。此外,研究者对HFIGs进行了基因本体论(GO)和京都基因与基因组百科全书(KEGG)功能富集分析。然后,对HFIGs进行了蛋白质相互作用(PPI)网络,并从PPI网络中确定了hub基因。此外,还进行了免疫浸润分析,以确定hub基因与免疫细胞浸润之间的相关性。为了验证GSE84044表达谱数据分析的可靠性,构建了CCl4诱导的HF大鼠模型,随后对HF大鼠和正常大鼠肝脏组织进行了转录组测序和免疫荧光分析及定量反转录(q-PCR)实验。最后,利用CMAP平台探索基于免疫靶点的HF治疗方法。结果显示,在GSE84044数据的生物信息学分析中,筛选出98个HFIGs,这些基因主要涉及炎症相关的生物途径,如NOD样受体信号通路、NF-kappa B信号通路、Toll样受体信号通路和PI3K-Akt信号通路。从PPI网络中,发现了10个hub基因,包括CXCL8、IL18、CXCL10、CD8A、IL7、PTPRC、CCL5、IL7R、CXCL9和CCL2。免疫浸润分析显示,免疫细胞如中性粒细胞、自然杀伤(NK)细胞、巨噬细胞M1和巨噬细胞M2与肝纤维化过程显著相关,hub基因表达与这些免疫细胞显著相关。值得注意的是,HFIGs富集的大多数生物途径和除CXCL8外的所有hub基因表达都在随后的转录组和qRCR实验中得到了验证。最后,筛选出15个有可能逆转hub基因高表达的小分子化合物作为HF的潜在治疗药物。综上,免疫基因CXCL8、IL18、CXCL10、CD8A、IL7、PTPRC、CCL5、IL7R、CXCL9和CCL2可能在HF的纤维化形成和炎症损害中发挥重要作用。这项研究的成果为研究HF的免疫机制提供了基础,有助于临床实践中对HF的诊断和预防及治疗。
方法解读
1. 从GEO数据库下载基因表达谱数据集GSE84044,包含124例肝纤维化患者的基因表达数据和肝组织炎症分级及纤维化分期等临床信息。
2. 使用WGCNA方法分析GSE84044数据集,设置阈值过滤基因和样本,构建基因共表达网络。计算每个模块与临床特征的相关性,获得与肝纤维化和炎症显著相关的基因模块。计算每个模块的模块成员度和基因显著性,获得与肝纤维化和炎症均显著相关的关键模块基因。
3. 从ImmPort数据库获得免疫相关基因,与WGCNA识别的关键模块基因比较,筛选肝纤维化免疫基因(HFIGs)。
4. 对HFIGs进行GO和KEGG功能富集分析。
5. 通过STRING和Cytoscape构建HFIGs的PPI网络,识别核心基因。
6. 使用CIBERSORT数据库分析GSE84044数据集免疫细胞浸润情况。
7. 建立四氯化碳诱导的大鼠肝纤维化模型,进行组织学、免疫荧光、转录组和qPCR实验验证分析结果。
8. 使用CMAP数据库筛选可能逆转核心基因表达的小分子化合物。
结果分析
1 WGCNA分析和HFIGs鉴定
研究者从GEO数据库下载HF的表达数据集,预处理后,对GSE84044数据进行了WGCNA分析。选择最佳软阈值β为5。然后,基于DynamicTreeCut算法,生成基因模块。如图1C所示,最终产生了21个基因模块。绘制Eigengene树状图和Eigengene邻接图来分析模块Eigengene(ME)的连接性,结果显示模块之间的距离大于0.25。见图1。研究者计算了模块-特征相关系数。结果显示,包括brown4, black, floralwhite, darkturquoise和red在内的5个模块与纤维化和炎症高度相关。因此,研究者选择了与纤维化和炎症显著相关的5个模块进行进一步分析。根据|GS|>0.4和|MM|>0.5以及p<0.05的标准,从5个重要模块中共确定了684个与纤维化和炎症高度相关的关键基因(图2,3)。最后,研究者比较从ImmPort数据库获得的2483个免疫基因,共筛选出98个IRG与纤维化和炎症相关的关键模块基因之间的重叠基因,并确定为肝纤维化免疫基因(HFIGs)(图4)。

图1.GSE84044数据的WGCNA。(A) 尺度独立性作为软阈值power的函数。(B) 平均连通性与软阈值power的关系。(C) 聚类树状图。每种颜色代表一个特定的共表达模块。聚类树下面的双色行代表原始模块和合并后的模块。(D) Eigengene树状图和模块eigengene邻接(MEs)。(E)MEs和临床性状之间的相关性热图。

图2.与炎症性状相关的模块的散点图分析。(A-E) 分别是红色、深绿色、黑色、棕色4和花白色的模块的散点图分析。
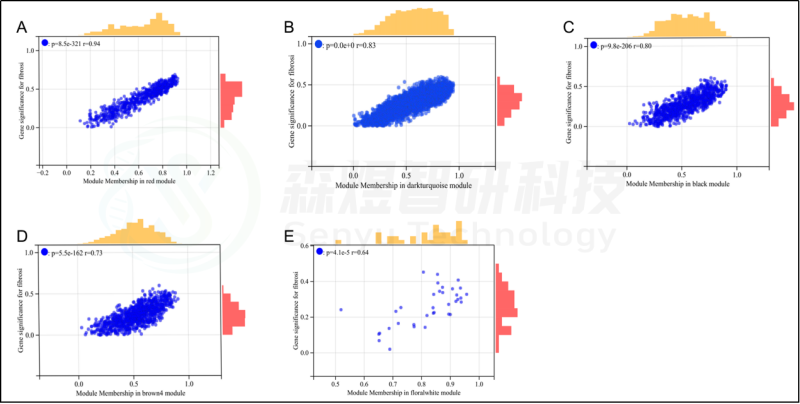
图3.与纤维化性状相关的模块的散点图分析。(A-E) 红色、深绿色、黑色、棕色4和花白色模块的散点图分析。
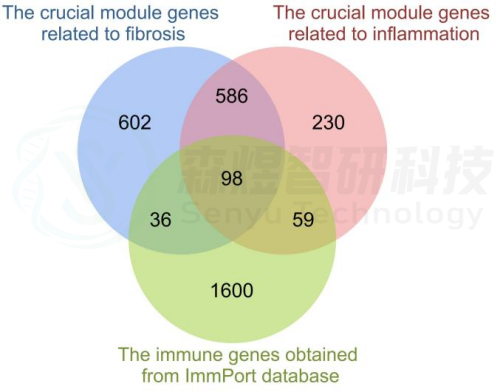
图4.HFIGs鉴定。
2 HFIGs的功能富集分析
为了确定HFIGs的生物学功能,对98个HFIGs进行功能富集分析,包括GO注释和KEGG途径富集分析。GO注释结果显示,98个HFIGs在149个生物过程(BP)、32个细胞成分(CC)和39个分子功能(MF)中显著富集。BP 涉及免疫反应、趋化性、中性粒细胞趋化性、炎症反应、信号转导、趋化因子信号通路、适应性免疫反应、抗原处理和呈递、杀死其他生物体的细胞等。CC包括质膜外侧、细胞外空间、细胞表面、ER至高尔基体运输囊膜、质膜的整体成分等。在MF中,98个HFIGs主要富集在趋化因子活性、肽抗原结合、CCR趋化因子受体结合、MHC II类受体活性、细胞因子活性、CXCR趋化因子受体结合、MHC II类蛋白复合物结合、T细胞受体结合等等。KEGG通路涉及:细胞因子-细胞因子受体相互作用、Th17细胞分化、抗原处理和表达、趋化因子信号通路、TNF信号通路、NF-kappa B信号通路、Toll样受体信号通路等。大多数富集的通路是与免疫有关的通路。见图5。
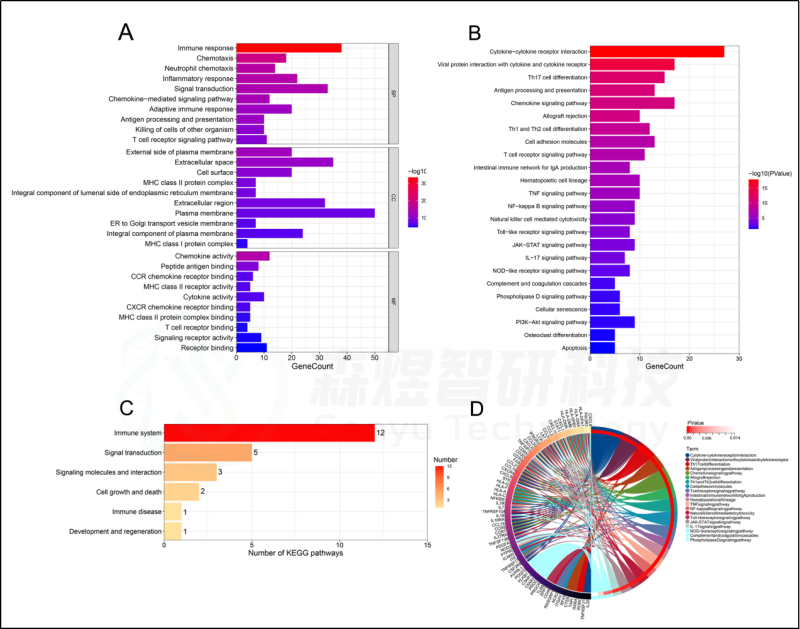
图5.HFIGs的功能富集分析。HFIGs的(A) GO注释。(B) KEGG途径富集分析。(C)KEGG信号通路的分类。(D)KEGG信号通路及其相关的HFIGs的相互作用。
3 PPI网络的构建与hub基因的选择
研究者构建了PPI网络。根据MCC算法,从PPI网络中筛选出排名前10个hub基因,并依次排序如下: CXCL8、IL18、CXCL10、CD8A、IL7、PTPRC、CCL5、IL7R、CXCL9和CCL2。这10个hub基因的表达与纤维化阶段(S0-S4)和炎症等级(G0-G4)呈正相关。所有的hub基因的表达都相互间有显著的相关性。见图6。功能富集分析显示,hub基因富集于多种KEGG途径。与98个HFIGs的KEGG通路富集结果相比,发现了8条共同的信号通路,包括细胞因子-细胞因子受体相互作用、病毒蛋白与细胞因子和细胞因子受体相互作用、趋化因子信号通路、Toll样受体信号通路、NOD样受体信号通路、IL-17信号通路、造血细胞系和TNF信号通路。见图6。

图6.PPI网络构建与hub基因选择。(A) 构建的PPI网络。(B) 10个从PPI网络中鉴定出的hub基因。(C) hub基因在不同纤维化阶段的肝组织中的表达。(D) 不同炎症级别的肝组织中hub基因的表达。(E) hub基因表达的相关性分析。(F) hub基因的功能富集分析。
4 免疫浸润分析
在肝脏组织中发现了21种浸润的免疫细胞类型,除了嗜酸细胞,它们在每个样本中都没有表达。随后,研究者评估了这些免疫细胞群之间的相关性。见图7。研究者还评估了每位患者肝脏组织中21种免疫细胞的浸润水平与纤维化阶段(S0-S4)和炎症等级(G0-G4)之间的相关性。纤维化阶段(S0-S4)与巨噬细胞M1、T细胞gamma delta和B细胞naive的浸润状态呈正相关,与B细胞记忆、巨噬细胞M2、NK细胞静止、中性粒细胞、树突状细胞激活和T细胞调节(Tregs)的浸润状态呈负相关。炎症等级(G0-G4)与T细胞γδ、巨噬细胞M1和B细胞幼稚期呈正相关,而与B细胞记忆期、巨噬细胞M2、NK细胞静止期、肥大细胞静止期、T细胞调节期(Tregs)和中性粒细胞呈负相关。见图8。此外,研究者进一步研究了10个hub基因与21种免疫细胞浸润的相关性。每个hub基因的表达与巨噬细胞M1、T细胞γδ和B细胞的浸润状态呈正相关,而与巨噬细胞M2、B细胞记忆、T细胞调节(Tregs)和NK细胞静止的浸润状态呈负相关。见图9。

图7.GSE84044数据的免疫浸润分析。(A)129例肝纤维化患者肝组织中免疫细胞浸润的热图。(B)129例肝纤维化患者肝组织中免疫细胞浸润的柱状图。(C)在22种不同的免疫细胞群之间的相关性。
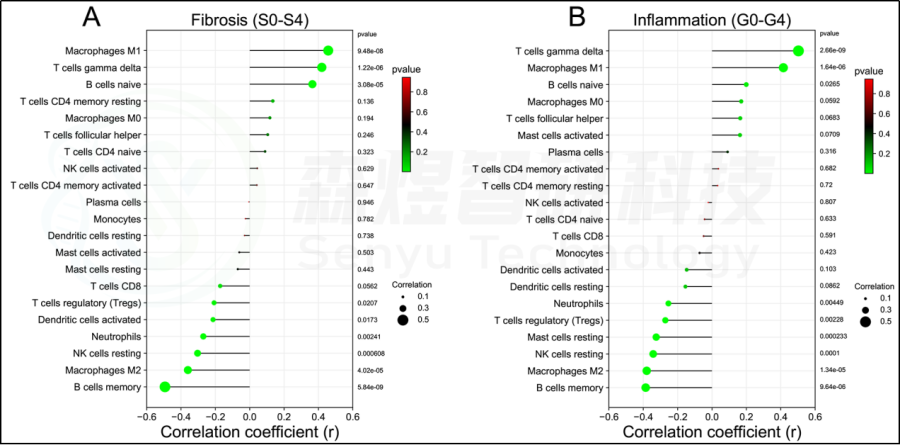
图8.HF患者肝组织纤维化分期与炎症分级与免疫细胞浸润的关系。(A)肝纤维化分期(S0-S4)。(B)肝脏炎症分级(G0-G4)。
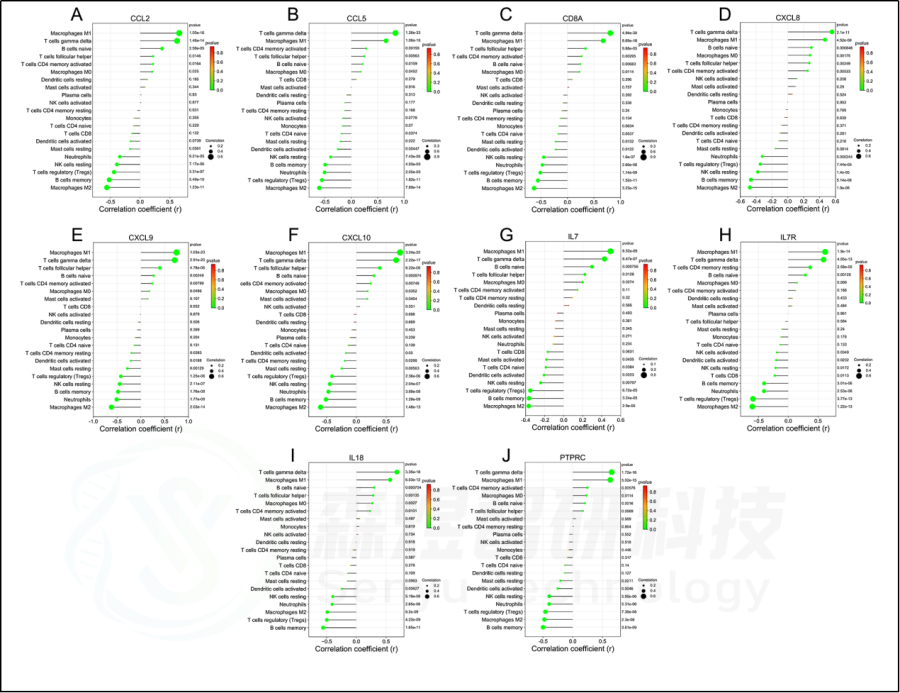
图9.肝纤维化患者肝组织中hub基因与免疫浸润的关系。(A) CCL2。(B) CCL5。(C) CD8A。(D) CXCL8。(E) CXCL8。(F) CXCL8。(G) IL7。(H) IL7R。(I) IL18。(J) PTPRC。
5 肝纤维化大鼠模型的建立
为了验证WGCNA分析结果的可靠性,研究者构建了一个HF的大鼠模型。结果显示,模型组大鼠的肝脏组织表现出大量的肝细胞脂肪变性、假小球的形成、肝细胞肿胀和坏死、免疫细胞的浸润和胶原纤维的广泛分布(图10)。ELISA结果显示,与正常对照组相比,CCl4模型组的肝脏HA、LN、IL1β、IL6和TNF-a均显著增加(图11)。

图10.苏木精和伊红(HE)染色和Masson染色。(A) HE染色。(B) Masson染色。
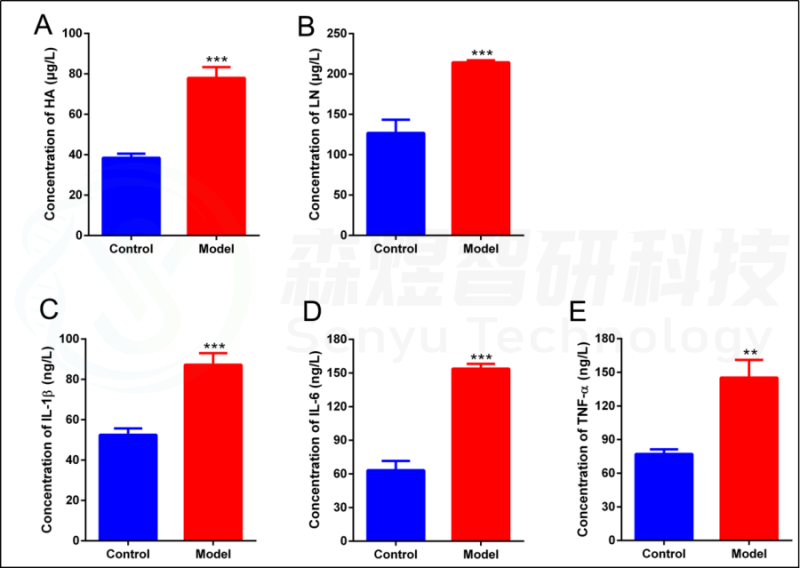
图11.采用ELISA法测定肝组织纤维化及炎症参数。(A) 透明质酸(HA)。(B) 层粘连蛋白(LN)。(C) 白细胞介素1b(IL1b)。(D) 白细胞介素6(IL6)。(E) 肿瘤坏死因子-a(TNF-a)。**p值<0.01;***p-value<0.001,与正常对照组比较。
6 免疫荧光染色
研究者对正常组和模型组的肝组织进行免疫荧光分析。结果显示,Cd86(巨噬细胞M1的标志物)在纤维化大鼠肝脏中的表达水平明显高于正常大鼠肝脏组织,但与正常对照组相比,模型组的Cd10(中性粒细胞的标志物)、Cd11b(NK细胞的标志物)和Cd206(巨噬细胞M2的标志物)表达水平明显下降。免疫荧光染色的结果在一定程度上验证了免疫浸润分析结果的准确性。见图12。
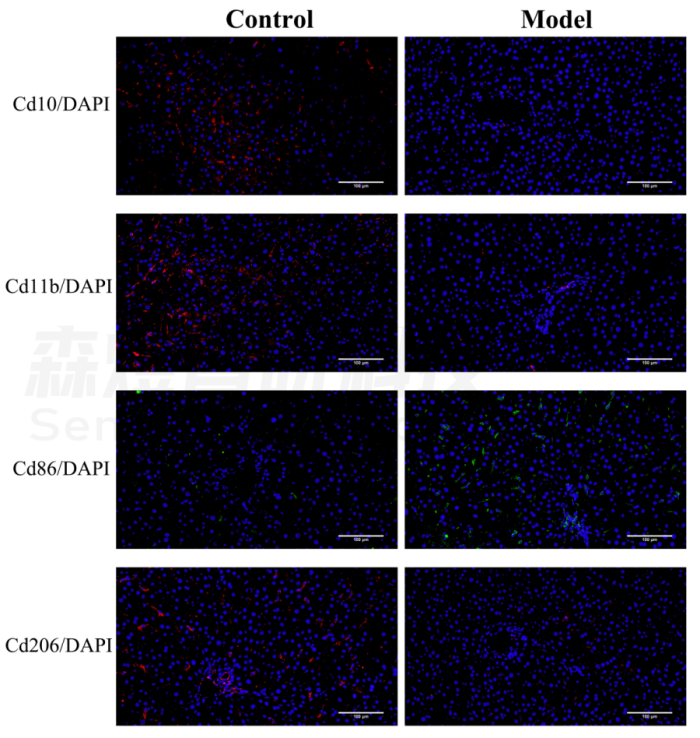
图12.免疫荧光染色检测免疫细胞浸润。
7 RNA-seq分析
研究者利用高通量测序技术对正常大鼠和CCl4诱导的肝纤维化大鼠的肝脏组织进行了检测,然后对差异基因进行筛选。筛选出1039个被注释的差异基因。主要富集在14条KEGG通路中,这些通路在之前的研究结果中被确定为HFIGs相关的KEGG信号通路,包括破骨细胞分化、趋化因子信号通路、自然杀伤细胞介导的细胞毒性、补体和凝血级联、Toll样受体信号通路、PI3K-Akt信号通路、NOD样受体信号通路、凋亡、NF-κB信号通路等。此外,研究者利用GSEA研究了在模型组和正常对照组中显著差异表达的信号通路。与前述结果中富含HFIGs的24条通路相比,研究者发现了19条共同的KEGG通路。如表1所示,与正常对照组相比,所有的共同途径在模型组都有显著的高表达。
研究者从RNA-seq数据中筛选出9个hub基因,包括Il18、Cxcl10、Cd8a、IL7、Ptprc、Ccl5、Il7r、Cxcl9和Ccl2(Cxcl8在大鼠物种中没有表达,所以不检测)。与正常肝脏组织相比,CCl4诱导的肝纤维化大鼠肝脏组织中的9个hub基因显著上调。Person相关分析显示所有9个hub基因之间有良好的相关性。总的来说,研究者的转录组分析结果进一步证实了这些免疫相关信号通路和hub基因在HF进展中的关键作用。见图13。

图13.RNA-seq数据的生物信息学分析。(A) RNA-seq数据中差异表达基因(DEGs)的热图。(B) RNA-seq数据中hub基因表达的相关性分析。(C) RNA-seq数据中正常组和CCl4模型组之间hub基因表达的变化。(D) 通过DAVID数据库富集的HFIGs相关和DEGs相关的共同信号通路。*p-value<0.05;**p-value<0.01;***p-value<0.001,表示功能富集结果的意义。
表1.GSEA富集的DEGs与DAVID数据库富集的HFIGs之间的共同信号通路。
ID | Description | Enrichment Score | NES | pvalue | qvalue |
ko04621 | NOD-like receptor signaling pathway | 0.52 | 1.72 | 1.07E-03 | 7.94E-03 |
ko04062 | Chemokine signaling pathway | 0.58 | 1.87 | 1.09E-03 | 7.94E-03 |
ko04060 | Cytokine-cytokine receptor interaction | 0.57 | 1.83 | 1.10E-03 | 7.94E-03 |
ko04514 | Cell adhesion molecules | 0.58 | 1.88 | 1.10E-03 | 7.94E-03 |
ko04380 | Osteoclast differentiation | 0.65 | 2.08 | 1.12E-03 | 7.94E-03 |
ko04064 | NF-kappa B signaling pathway | 0.60 | 1.87 | 1.14E-03 | 7.94E-03 |
ko04612 | Antigen processing and presentation | 0.61 | 1.88 | 1.14E-03 | 7.94E-03 |
ko04650 | Natural killer cell mediated cytotoxicity | 0.75 | 2.32 | 1.15E-03 | 7.94E-03 |
ko04658 | Th1 and Th2 cell differentiation | 0.63 | 1.91 | 1.19E-03 | 7.94E-03 |
ko04640 | Hematopoietic cell lineage | 0.73 | 2.22 | 1.19E-03 | 7.94E-03 |
ko05330 | Allograft rejection | 0.76 | 2.16 | 1.25E-03 | 7.94E-03 |
ko04061 | Viral protein interaction with cytokine and cytokine receptor | 0.69 | 1.92 | 1.27E-03 | 7.94E-03 |
ko04672 | Intestinal immune network for IgA production | 0.81 | 2.15 | 1.31E-03 | 7.94E-03 |
ko04072 | Phospholipase D signaling pathway | 0.55 | 1.76 | 2.20E-03 | 1.19E-02 |
ko04620 | Toll-like receptor signaling pathway | 0.52 | 1.62 | 4.55E-03 | 1.89E-02 |
ko04659 | Th17 cell differentiation | 0.50 | 1.57 | 6.77E-03 | 2.50E-02 |
ko04660 | T cell receptor signaling pathway | 0.48 | 1.49 | 1.26E-02 | 4.17E-02 |
ko04151 | PI3K-Akt signaling pathway | 0.39 | 1.33 | 1.64E-02 | 5.02E-02 |
ko04610 | Complement and coagulation cascades | 0.44 | 1.38 | 3.64E-02 | 9.01E-02 |
8 通过q-PCR验证hub基因的表达
由于Cxcl8在大鼠体内不表达,因此,研究者通过q-PCR实验检测了包括Il18、Cxcl10、Cd8a、IL7、Ptprc、Ccl5、Il7r、Cxcl9和Ccl2在内的9个hub基因的mRNA表达水平,以验证正常组和模型组肝组织中确定的hub基因的表达。结果显示,与正常对照组相比,模型组中hub基因的mRNA表达显著上调,与之前的WGCNA分析和RNA测序结果一致。

图14.通过q-PCR检测正常组和四氯化碳模型组中9个hub基因的mRNA表达量。*p-value< 0.05;**p-value<0.01;***p-value<0.001,与正常对照组比较。
9 候选小分子的鉴定
研究者通过CMAP筛选逆转HF的潜在药物。筛选出连通性分数为负的前15个小分子化合物,包括elotristat, lomitapide, malotilate, tranylcypromine, TG-101348, cordycepin, tafamidis-meglumine, fostamatinib, delcorine, phenylbutazone, endo-IWR-1, scopolamine, anidulafungin, simvastatin 和 terconazole(表2)。这些小分子化合物有可能通过抑制hub基因的上调而发挥抗肝纤维化的作用。
表2.小分子鉴定结果。
Rank | CMAP name | Connectivity score | -log (p value) |
1 | telotristat | -0.912 | 15.6536 |
2 | lomitapide | -0.883 | 15.6536 |
3 | malotilate | -0.8735 | 15.6536 |
4 | tranylcypromine | -0.8728 | 15.6536 |
5 | TG-101348 | -0.8707 | 15.6536 |
6 | cordycepin | -0.8564 | 15.6536 |
7 | tafamidis-meglumine | -0.8454 | 12.2035 |
8 | fostamatinib | -0.8445 | 11.9793 |
9 | delcorine | -0.8442 | 2.9231 |
10 | phenylbutazone | -0.8437 | 2.8391 |
11 | endo-IWR-1 | -0.8432 | 2.7737 |
12 | scopolamine | -0.8392 | 2.4983 |
13 | anidulafungin | -0.8386 | 1.9741 |
14 | simvastatin | -0.8371 | 1.9178 |
15 | terconazole | -0.835 | 1.8569 |
结论
本研究表明,免疫机制在HF的发生和发展中起着关键作用,并探索了基于免疫靶点的HF治疗方法。研究者筛选了98个与HF相关的免疫基因,并从中确定了10个hub基因,包括CXCL8、IL18、CXCL10、CD8A、IL7、PTPRC、CCL5、IL7R、CXCL9和CCL2。研究者在HF大鼠的肝脏组织中检测了这9个hub基因的表达,结果表明这些hub基因可能对HF的纤维化形成和炎症性损伤有贡献。研究者确定了几个与HF相关的免疫信号通路,如NOD样受体信号通路、NF-kappa B信号通路、Tolllike受体信号通路、PI3K-Akt信号通路、T细胞受体信号通路和磷脂酶D信号通路。免疫浸润分析和免疫荧光分析表明,免疫细胞如中性粒细胞、NK细胞、巨噬细胞M1和巨噬细胞M2的浸润状况与HF显著相关。综合来看,本研究揭示了HF的潜在免疫机制,并研究了逆转HF的免疫治疗药物,该疾病的具体分子机制和小分子药物的药理机制还需要进一步探索。
原名:Revealing immune infiltrate characteristics and potential immune-related genes in hepatic fibrosis: based on bioinformatics,transcriptomics and q-PCR experiments
译名:基于生物信息学、转录组学和q-PCR实验的结果揭示肝纤维化中的免疫浸润特征和潜在的免疫相关基因
期刊:Frontiers in Immunology
IF:8.786
发表时间:2023年4月
DOI号:10.3389/fimmu.2023.1133543



