背景介绍
本研究主要使用不同的生化和生物物理方法检验了玫瑰红嗜热球菌 N-去甲基化酶与嗜温枯草芽孢杆菌的 N-脱甲基酶的高热稳定性的性质,有助于理解嗜热酶的天然稳定框架,为工业应用中构建稳健酶提供参考。去甲基化是许多天然产物转化为活性形态的关键步骤,但是-C-N-键极为稳定,氮原子含有未成键的孤电子对,环上电子云密度大,碳氮键键长短,难以极化,且相关反应效率低,使得含氮基团较难去除,因此N-甲基脱除反应是目前有机合成的一大挑战。在团队前期研究工作中发现了一种来自玫瑰红嗜热球菌(Thermomicrobium roseum DSM 5159)的N-甲基脱除酶,即肌氨酸氧化酶(Sarcosine oxidase, EC 1.5.3.1, TrSOX),属于黄素蛋白氧化酶类,以FAD为辅因子,该酶针对N-甲基具有高效的生物脱除能力,并且具有一定的手性选择性,表现出优异的耐热性和环境抗性。
设计思路
该研究指出几个影响酶热稳定性的关键因素,包括二硫键、盐桥、疏水性、氢键、螺旋含量和柔韧性,以及含有脯氨酸等刚性侧链的残基被认为在酶的稳定性中起着关键作用。此外,在蛋白质或基于蛋白质的药物产品的开发中,聚集或聚集相关的沉淀一直被认为是主要挑战之一,它会导致生物功能的丧失。
图文导读
该研究基于玫瑰红嗜热球菌N-甲基脱除酶TrSOX的晶体结构,在聚集界面和刚性位点上引入取代,以降低聚集比和刚性。在聚集界面上,V162S、M308S、F170S和V306S的4个取代热稳定性显著降低,但催化效率有一定程度的提升。此外,耐热性框架在几个多重P→G的取代(P129G/P134G, P237G/P259G和P259G/P276G)中被严重破坏。这些结构波动与分子动力学模拟中的整体结构和局部的RMSD、氢键旋转半径和溶剂可溶表面积值具有良好的一致性。为了进一步确认TrSOX上聚集界面和脯氨酸残基的作用,将这些关键位点引入枯草芽孢来源的肌氨酸氧化酶,这些取代提高了Tm和ΔH值,氢键数增加,降低了RSMD、Rg和SASA值,促进热稳定性。尤其是突变体A249P、F309V、E161V和N238P,它们在60°C下的半衰期从小于10 min显著增加到1440 min、996min、640min和60min。

Figure 1. Structural design. (A) Aggregation interface of TrSOX. (B) The substitutions were designed to reduce the aggregation ratio on the aggregation interface (marked in yellow). (C) Flexible substitutions were designed at the surface proline sites (marked in yellow). (D) Structural and sequential alignments of TrSOX (green) and BSOX (white), and they shared ∼36.6% sequence identity.

Figure 2. The RMSD values of substitutions on the aggregation interface. Whole-structure RMSD values of substitutions at (A) 300 K and (B) 350K. Substrate pocket RMSD of substitutions at(C)300K and(D)350K.

Figure 3. RMSD values of substitutions at proline sites. Whole-structure RMSD values of substitutions at (A) 300 K and (B) 350 K. Substrate pocket RMSD values of substitutions at (C) 300 K and (D) 350 K.

Figure 4. RMSD values of S320K/P129G/P134G. Whole structure RMSD values of substitutions at (A) 300 K and (B) 350 K. Partial RMSD (129−134) RMSD values of substitutions at (C) 300 K and (D) 350 K. Substrate pocket RMSD values of substitutions at (E) 300 K and (F) 350K. (G) Location of the fragment (129−134, marked in yellow).


Figure 5. RMSD values of S320K/P237G/P259G. Whole-structure RMSD values of substitutions at (A) 300 K and (B) 350 K. Partial RMSD (237−259) values of substitutions at (C) 300 K and (D) 350 K. Substrate pocket RMSD values of substitutions at (E) 300 K and (F) 350 K. (G) Location of the fragment (129−134, marked in yellow; 237−259, marked in green).
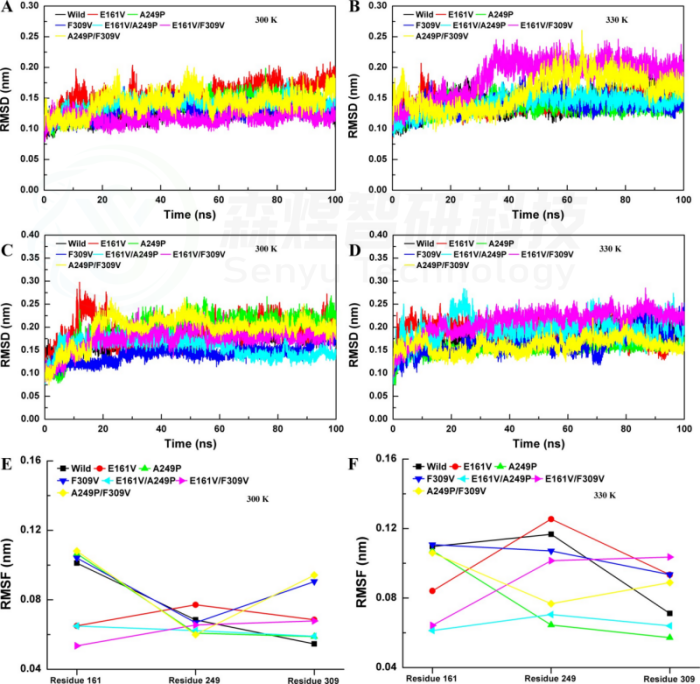
Figure 6. RMSD values of S320K/P259G/P276G. Whole-structure RMSD values of substitutions at (A) 300 K and (B) 350 K. Partial RMSD (259−276) values of substitutions at (C) 300 K and (D) 350 K. Substrate pocket RMSD values of substitutions at (E) 300 K and (F) 350 K. (G) (129−134, marked in yellow; 237−259, marked in green; 259−276, marked in pink).
文献档案
原名:Aggregation Interface and Rigid Spots Sustain the Stable Framework of a Thermophilic
N-Demethylase
期刊:Journal of Agricultural and Food Chemistry
DOI号: 10.1021/acs.jafc.3c00877



